研究開発本部の山本です。前回は、フリーで利用可能なメタボロームデータ、トランスクリプトームデータをご紹介しました。今回はこれらのオミックスデータに対し、フリーの解析サービスであるMetaboAnalystを用いて主成分分析を行う具体的な手順についてご紹介します。
主成分分析は、メタボロームデータの統計解析手法として最も良く用いられる手法で、メタボローム解析の論文を読めば、必ずどこかで出てくると言ってよいほど、広く用いられています。メタボロームデータに主成分分析を行った結果をどのように利用し、生物学的な解釈に繋げていくのかは次回以降説明することとして、今回はMetaboAnalystを用いて主成分分析を実行してみましょう。
MetaboAnalystについて簡単にご紹介します。MetaboAnalystは、カナダのアルバータ大学のDavid Wishartと、マギル大学のJeff Xiaを中心に運営されているフリーのWebサービスで、メタボロームデータの統計解析に特化して作られています。MetaboAnalystの詳細は彼らの論文に詳しいです。
アルバータ大学のDavid Wishartのグループと言えば、ヒトのメタボロームデータベースであるHuman metabolome database(HMDB)で馴染みのある方もいらっしゃるかもしれませんが、MetaboAnalystもHMDBを作っているグループによって開発、運営されています。
では実際にMetaboAnalystを用いてメタボロームデータに主成分分析を適用してみましょう。
MetaboAnalystで主成分分析を行うためのデータの準備として、各行に代謝物質、各列にサンプルの代謝物質×サンプルの行列データを準備します。
前回ご紹介した慶應義塾大学の大腸菌マルチオミックスデータベースにあるメタボロームデータは、代謝物質×サンプルの行列データになっていますので、少し整形するだけで主成分分析を適用することが可能です。
データの1行目をサンプル名(欠損遺伝子名、もしくは増殖速度)、2行目に群番号、1列名を代謝物質名とし、3行目から各代謝物質レベルのデータになるように整形します。群番号は主成分分析の実行には不要ですが、その他の手法を適用する際に必要となるので、データに加えておきます。主成分分析を行うだけであれば、全て同じ群名(例えば1)を入力しておけば十分です。ファイルはCSV形式で保存してください。
では実際に主成分分析を実行します。”Statistical Analysis”をクリックし、Uploadからデータをアップロードします。Formatは、各列にサンプルの情報がcsvファイルに書かれているので、”Samples in columns (unpaired)”としてSubmitを押します。
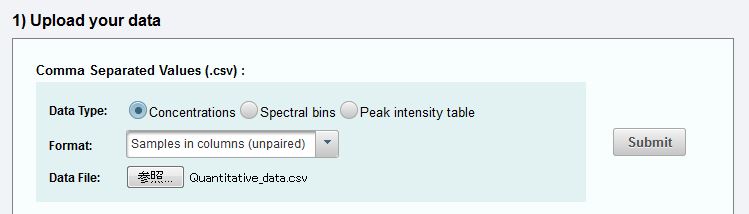
データのフォーマットが正しければ次に進むことが出来ますが、上記のデータのままではエラーが出るはずです。
エラーメッセージを読むと、”Citrateが重複している”旨のメッセージが表れます。改めてcsvファイルのデータを確認してみると、確かにCitrateと名前が付いた行が2つあり、1つは全てのサンプルが空白の行になっています。これは、ピークの解析の際に削除しきれなかったものが残っていたのではないか、と考えられます。そこで2つのCitrateのうち、全てのサンプルで空白になっている行を削除し、改めてデータのアップロードを行います。
 その後、様々設定を要求されますが、今回は何も考えずとりあえずSkipまたはSubmitを押してどんどん進んでください。
その後、様々設定を要求されますが、今回は何も考えずとりあえずSkipまたはSubmitを押してどんどん進んでください。
最後まで進むと、t検定などの様々な解析方法のリストが表示される「Select an analysis path to explore」という画面にたどり着きます。今回は主成分分析なので、”Multivariate Analysis”の”Principal Component Analysis(PCA)”をクリックすると、結果が表示されます。
メタボローム解析の論文でよく見るスコアプロットは、”2D Scores Plot”のタブをクリックすると表示されます。今回のデータでは、次のような第一主成分と第二主成分のスコアプロットが得られているはずです。

以上で、MetaboAnalystを用いてメタボロームデータに主成分分析を行うことができました。まだ主成分分析をご自身で行ったことのない方は、一度お試しいただければと思います。
次回は、今回いくつかスキップした設定や、主成分分析の結果をどのように見ればよいのか、因子負荷量の利用法等について、ご説明したいと思います。
